Capítulo 30 El paquete caret
Ya hemos aprendido sobre regresión y kNN como algoritmos machine learning y, en secciones posteriores, aprenderemos sobre varios otros. Todos estos son solo un pequeño subconjunto del total de los algoritmos disponibles. Muchos de estos algoritmos se implementan en R. Sin embargo, se distribuyen a través de diferentes paquetes, desarrollados por diferentes autores, y a menudo usan una sintaxis diferente. El paquete caret intenta consolidar estas diferencias y dar consistencia. Actualmente incluye 237 métodos diferentes que se resumen en el manual del paquete caret110. Tengan en cuenta que caret no incluye los paquetes necesarios y, para implementar un paquete a través de caret, tendrán que instalar el paquete. Los paquetes requeridos para cada método se describen en el manual del paquete.
El paquete caret también provee una función que realiza la validación cruzada para nosotros. Aquí oferecemos algunos ejemplos que muestran cómo utilizamos este paquete increíblemente útil. Usaremos el ejemplo “2 o 7” para ilustrar:
library(tidyverse)
library(dslabs)
data("mnist_27")30.1 La función train de caret
La función train de caret nos permite entrenar diferentes algoritmos utilizando una sintaxis similar. Entonces, por ejemplo, podemos escribir:
library(caret)
train_glm <- train(y ~ ., method = "glm", data = mnist_27$train)
train_knn <- train(y ~ ., method = "knn", data = mnist_27$train)Para hacer predicciones, podemos usar el resultado de esta función directamente sin necesidad de mirar los detalles de predict.glm y predict.knn. En cambio, podemos aprender cómo obtener predicciones de predict.train.
El código se ve igual para ambos métodos:
y_hat_glm <- predict(train_glm, mnist_27$test, type = "raw")
y_hat_knn <- predict(train_knn, mnist_27$test, type = "raw")Esto nos permite comparar rápidamente los algoritmos. Por ejemplo, podemos comparar la exactitud de esta manera:
confusionMatrix(y_hat_glm, mnist_27$test$y)$overall[["Accuracy"]]
#> [1] 0.75
confusionMatrix(y_hat_knn, mnist_27$test$y)$overall[["Accuracy"]]
#> [1] 0.8430.2 Validación cruzada
Cuando un algoritmo incluye parámetros de ajuste, train automáticamente utiliza la validación cruzada para decidir entre algunos valores predeterminados. Para saber qué parámetro o parámetros están optimizados, pueden leer el manual111 o estudiar lo que devuelve:
getModelInfo("knn")También podemos usar una búsqueda rápida como esta:
modelLookup("knn")Si lo ejecutamos con valores predeterminados:
train_knn <- train(y ~ ., method = "knn", data = mnist_27$train)pueden ver rápidamente los resultados de la validación cruzada utilizando la función ggplot. El argumento highlight destaca el máximo:
ggplot(train_knn, highlight = TRUE)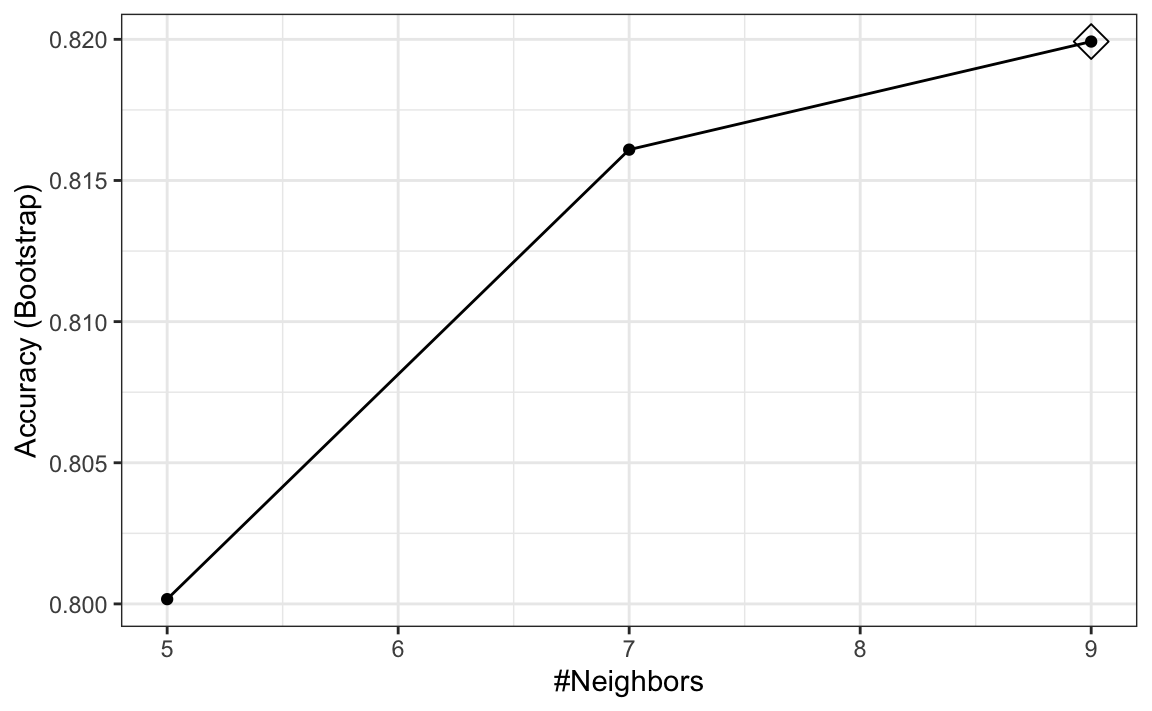
Por defecto, la validación cruzada se realiza tomando 25 muestras de bootstrap que comprenden el 25% de las observaciones. Para el método kNN, por defecto se intenta \(k=5, 7, 9\). Cambiamos esto usando el parámetro tuneGrid. La cuadrícula de valores debe ser suministrada por un data frame con los nombres de los parámetros como se especifica en el output de modelLookup.
Aquí, presentamos un ejemplo donde probamos 30 valores entre 9 y 67. Para hacer esto con caret, necesitamos definir una columna llamada k de la siguiente manera: data.frame(k = seq(9, 67, 2)).
Noten que al ejecutar este código, estamos ajustando 30 versiones de kNN a 25 muestras de bootstrap. Como estamos ajustando \(30 \times 25 = 750\) kNN modelos, ejecutar este código tomará varios segundos. Fijamos la semilla porque la validación cruzada es un procedimiento aleatorio y queremos asegurarnos de que el resultado aquí sea reproducible.
set.seed(2008)
train_knn <- train(y ~ ., method = "knn",
data = mnist_27$train,
tuneGrid = data.frame(k = seq(9, 71, 2)))
ggplot(train_knn, highlight = TRUE)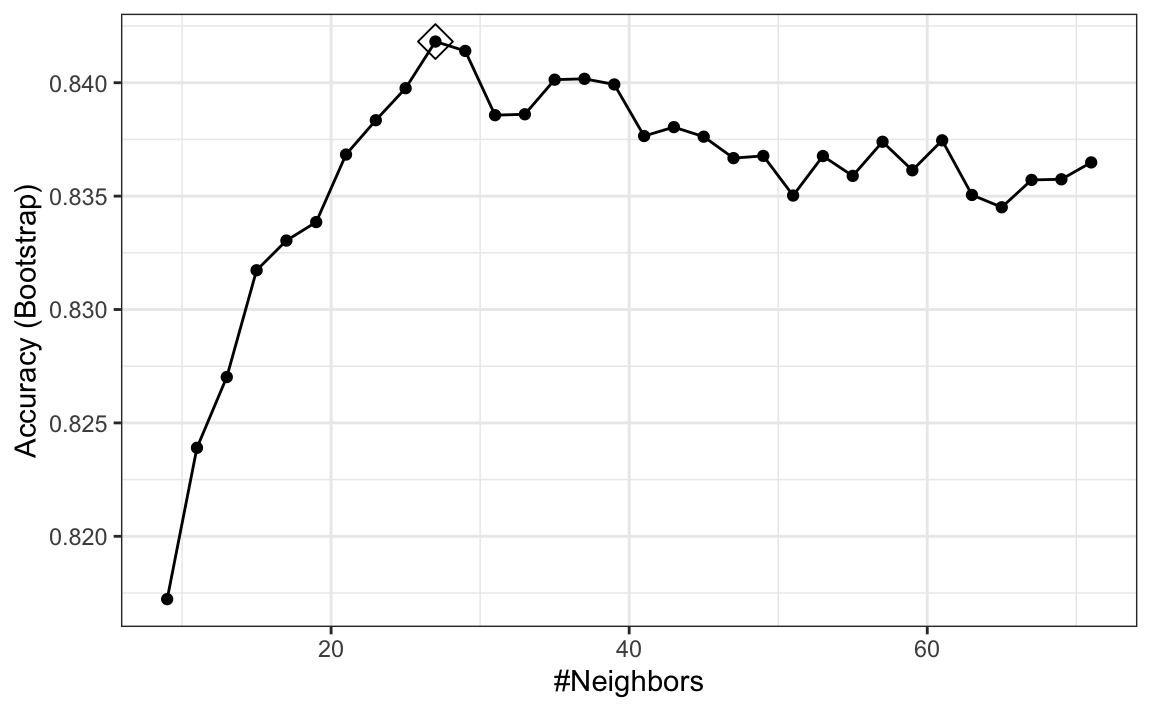
Para acceder al parámetro que maximiza la exactitud, pueden usar esto:
train_knn$bestTune
#> k
#> 10 27y para acceder el mejor modelo, pueden usar esto:
train_knn$finalModel
#> 27-nearest neighbor model
#> Training set outcome distribution:
#>
#> 2 7
#> 379 421La función predict utilizará este modelo de mejor rendimiento. Aquí está la exactitud del mejor modelo cuando se aplica al set de evaluación, que todavía no hemos utilizado porque la validación cruzada se realizó en el set de entrenamiento:
confusionMatrix(predict(train_knn, mnist_27$test, type = "raw"),
mnist_27$test$y)$overall["Accuracy"]
#> Accuracy
#> 0.835Si queremos cambiar la forma en que realizamos la validación cruzada, podemos usar la función trainControl. Podemos hacer que el código anterior sea un poco más rápido mediante, por ejemplo, la validación cruzada 10 fold. Esto significa que tenemos 10 muestras y cada una usa el 10% de las observaciones. Logramos esto usando el siguiente código:
control <- trainControl(method = "cv", number = 10, p = .9)
train_knn_cv <- train(y ~ ., method = "knn",
data = mnist_27$train,
tuneGrid = data.frame(k = seq(9, 71, 2)),
trControl = control)
ggplot(train_knn_cv, highlight = TRUE)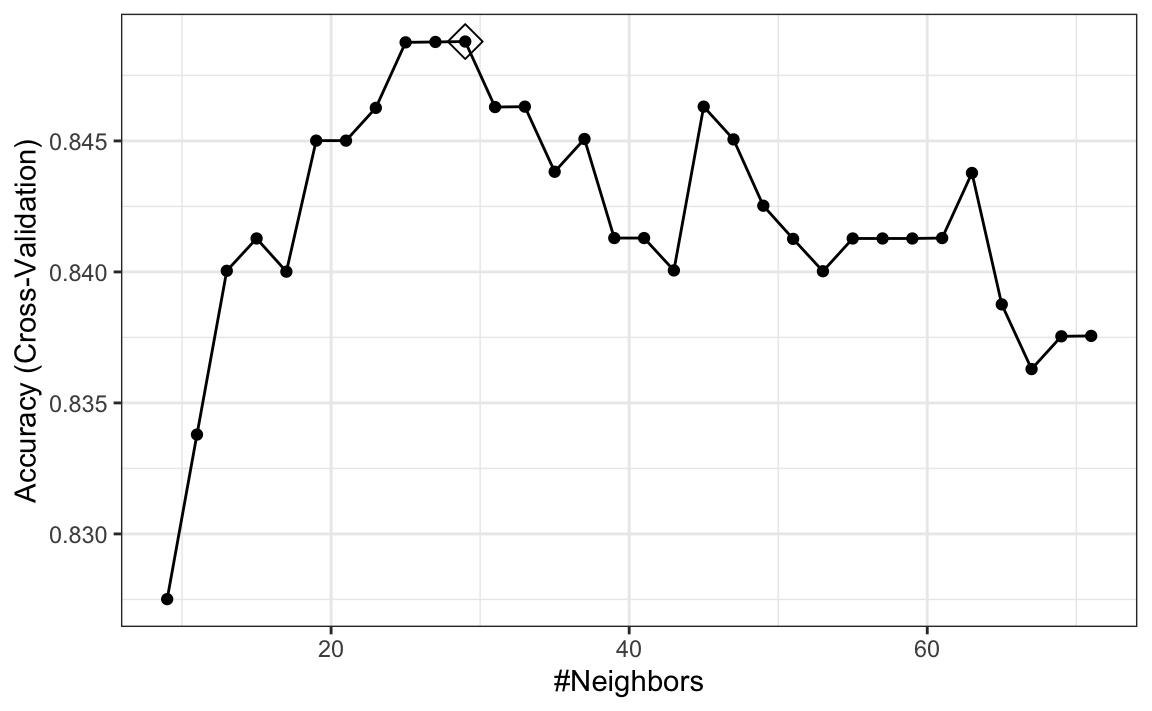
Observamos que los estimadores de exactitud son más variables, algo esperado ya que cambiamos el número de muestras utilizadas para estimar la exactitud.
Tengan en cuenta que el componente results de lo que devuelve train incluye varias estadísticas de resumen relacionadas con la variabilidad de los estimadores de validación cruzada:
names(train_knn$results)
#> [1] "k" "Accuracy" "Kappa" "AccuracySD" "KappaSD"30.3 Ejemplo: ajuste con loess
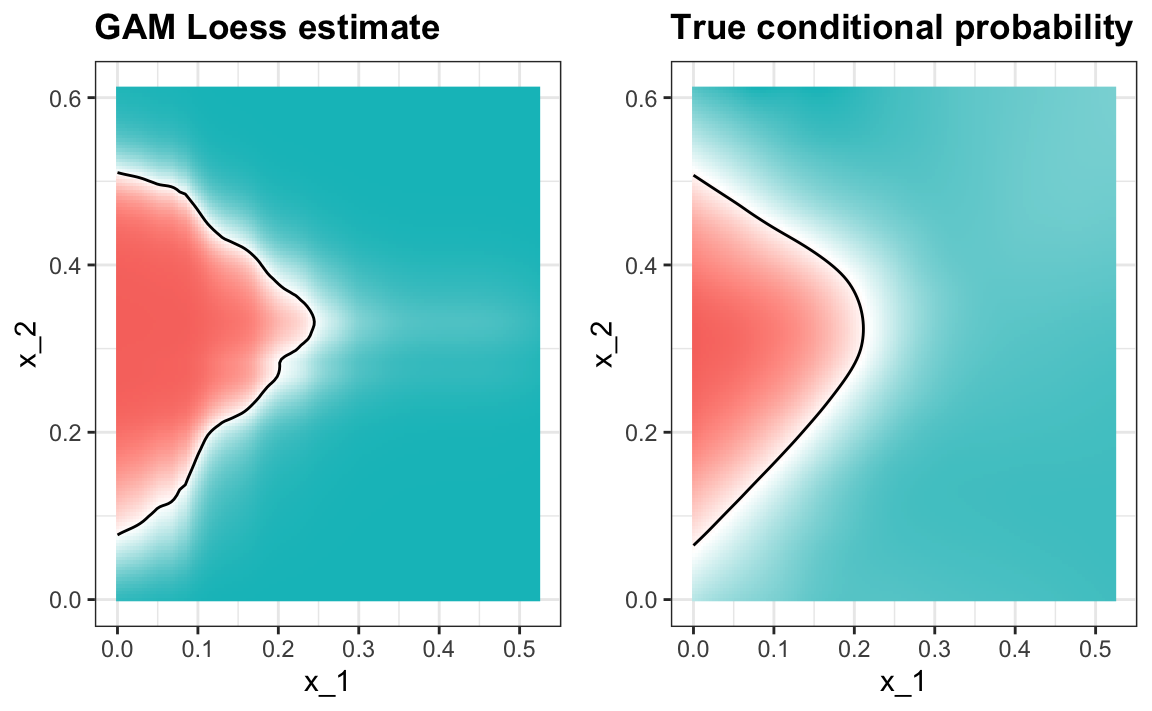
El modelo kNN que da el mejor resultado también da una buena aproximación de la probabilidad condicional verdadera:
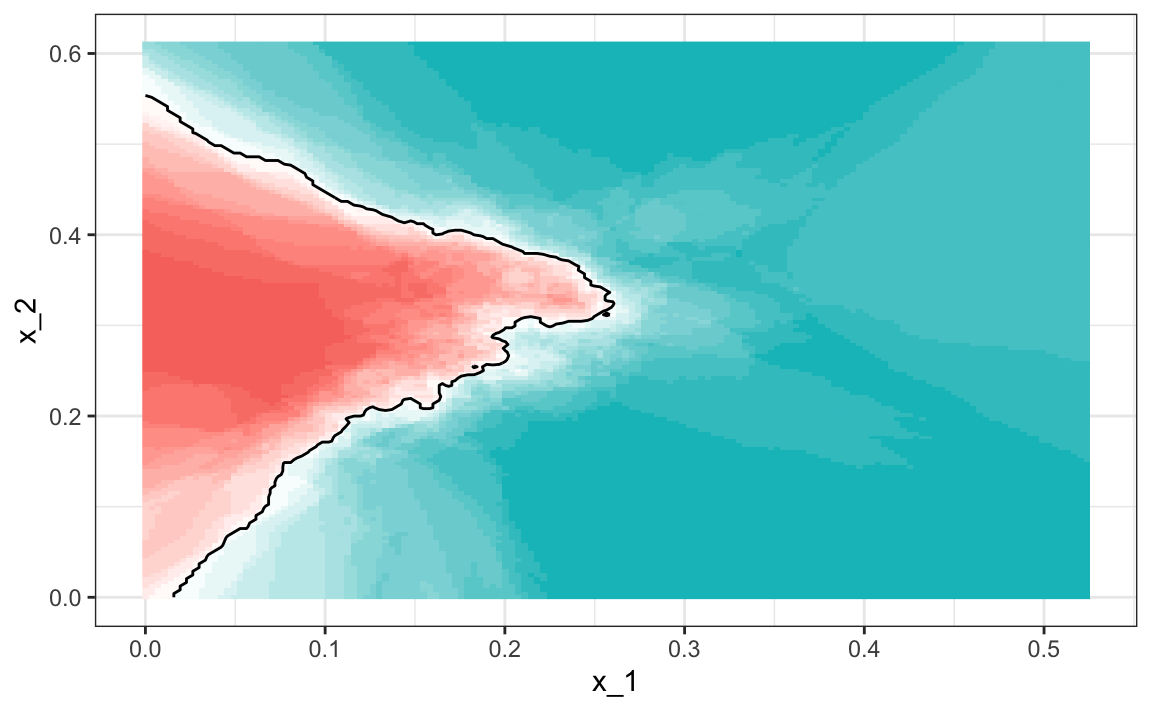
Sin embargo, sí vemos que la frontera es algo ondulada. Esto se debe a que kNN, igual que el suavizador de compartimiento básico, no utiliza un kernel. Para mejorar esto, podríamos tratar loess. Al leer la parte sobre los modelos disponibles en el manual112, vemos que podemos usar el método gamLoess.
En el manual113, también vemos que necesitamos instalar el paquete gam si aún no lo hemos hecho:
install.packages("gam")Luego, vemos que tenemos dos parámetros para optimizar:
modelLookup("gamLoess")
#> model parameter label forReg forClass probModel
#> 1 gamLoess span Span TRUE TRUE TRUE
#> 2 gamLoess degree Degree TRUE TRUE TRUENos mantendremos en un grado de 1. Pero para intentar diferentes valores para span, aún tenemos que incluir una columna en la tabla con el nombre degree:
grid <- expand.grid(span = seq(0.15, 0.65, len = 10), degree = 1)Utilizaremos los parámetros de control de validación cruzada predeterminados.
train_loess <- train(y ~ .,
method = "gamLoess",
tuneGrid=grid,
data = mnist_27$train)
ggplot(train_loess, highlight = TRUE)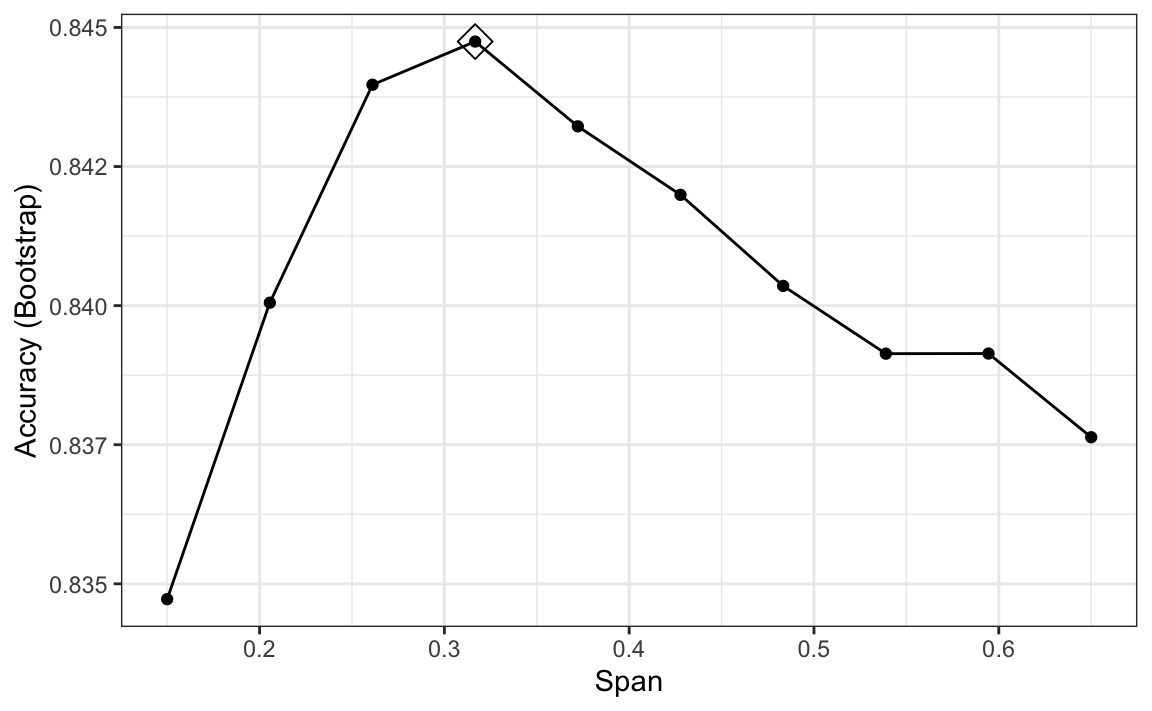
Podemos ver que el método funciona de manera similar a kNN:
confusionMatrix(data = predict(train_loess, mnist_27$test),
reference = mnist_27$test$y)$overall["Accuracy"]
#> Accuracy
#> 0.85y produce un estimador más suave de la probabilidad condicional: